「ゲノム編集」遺伝子を発見するソフトウェアを開発
- プレスリリース
2018年10月12日
1.発表者:
- 森 秀人
- (東京大学先端科学技術研究センター 交流研究生/慶應義塾大学政策・メディア研究科 博士課程1年)
- 谷内江 望
- (東京大学先端科学技術研究センター 合成生物学分野 准教授)
2.発表のポイント:
- 大規模ゲノムデータから周期的なリピート配列を高速探査するソフトウェアを開発しました。
- 近年ゲノムの配列を書き換える技術として発展している「ゲノム編集」技術に関連する遺伝子や、構造タンパク質遺伝子、微生物の進化に重要であると考えられる周期的なDNA、タンパク質アミノ酸配列を網羅的かつ正確に捕捉できるようになりました。
- 世界的にゲノム編集技術の知的財産競争も熾烈な中、新たなゲノム編集関連遺伝子の発見につながることが期待されます。
3.発表概要:
近年様々な生物種の染色体DNA配列を人工的に書き換えることができるゲノム編集技術の研究開発が世界的に進んでいます。東京大学先端科学技術研究センターの谷内江 望 准教授と慶應義塾大学先端生命科学研究所の冨田 勝 教授らによる研究グループは、CRISPR(注1)、ジンクフィンガーヌクレアーゼ(ZFN:注2)、TALEヌクレアーゼ(TALEN:注3)など全てのゲノム編集技術の基礎となった遺伝子が反復的なDNA配列やタンパク質アミノ酸配列を持つことに注目し、大規模ゲノムデータから多様なリピート配列を高速探査するソフトウェア「SPADE(Search for Patterned DNA Elements)」を開発しました。SPADEによって正確に既知のゲノム編集関連遺伝子、構造タンパク質遺伝子、微生物進化に重要であると考えられる周期的なDNA配列を網羅的かつ正確に捕捉できるようになりました。世界的にゲノム編集技術の知的財産競争も熾烈な中、本研究によって新たなゲノム編集関連遺伝子や新規機能をもった遺伝子の発見、ゲノム進化の新しいメカニズムの研究が加速することが期待されます。
本成果は10月10日付で「Nucleic Acids Research」に掲載されました。
4.発表内容:
近年ゲノム編集技術の開発が進み、様々な生物種において染色体DNA配列を人工的に書き換えることが可能になりつつあります。これらの技術は生物学を大きく推進することはもちろん、医療分野、産業分野への応用開発も進んでいます。一方で、世界中でこれらの技術に関わる知的財産を巡る争いも熾烈で、高性能で新たなタイプのゲノム編集技術を開発することも重要な課題です。
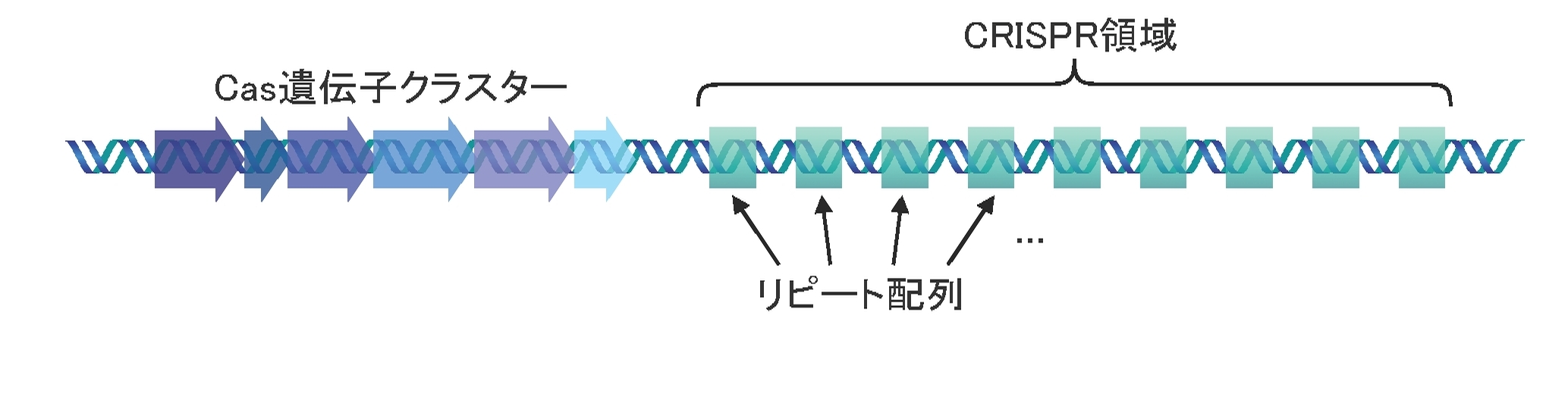
研究グループは、これまで実用化あるいは開発が進んでいるゲノム編集技術は全て自然界における生物が持つ規則的なDNA配列やタンパク質アミノ酸配列に由来することに着目しました。例えば、ジンクフィンガーヌクレアーゼ(ZFN)やTALEヌクレアーゼ(TALEN)というゲノム編集技術ではアミノ酸配列を部分的に変更することによって自在に任意のDNAに結合できるように改変できるタンパク質が利用されていますが、これらのタンパク質はDNAの二重らせん構造に巻き付くための周期的なアミノ酸配列を持っています(図1)。近年急速に利用が広がっているCRISPRも元来はバクテリアのウィルスに対する免疫獲得機構として発見されましたが、ウィルスDNA配列を免疫する際にバクテリアゲノムに一定間隔を空けた周期的な反復配列が形成されていくことが知られています(図2)。この他、タンパク質間相互作用、遺伝子発現、細胞周期、シグナル伝達など様々な生物学的プロセスの制御に関連しているタンパク質やクモ糸を形成するフィブロインといった様々な構造タンパク質は一定の周期をもつアミノ酸配列を持ちます。さらに、生物進化もゲノム配列中の周期的な配列が推進力として働いていることが示唆されています。このように、周期的リピート配列に注目することで、新規ゲノム編集技術につながる遺伝子や新しい生物メカニズムの発見が期待されるにも関わらず、これまでに周期的なリピート配列を高速かつ網羅的に同定する手法は開発されていませんでした。
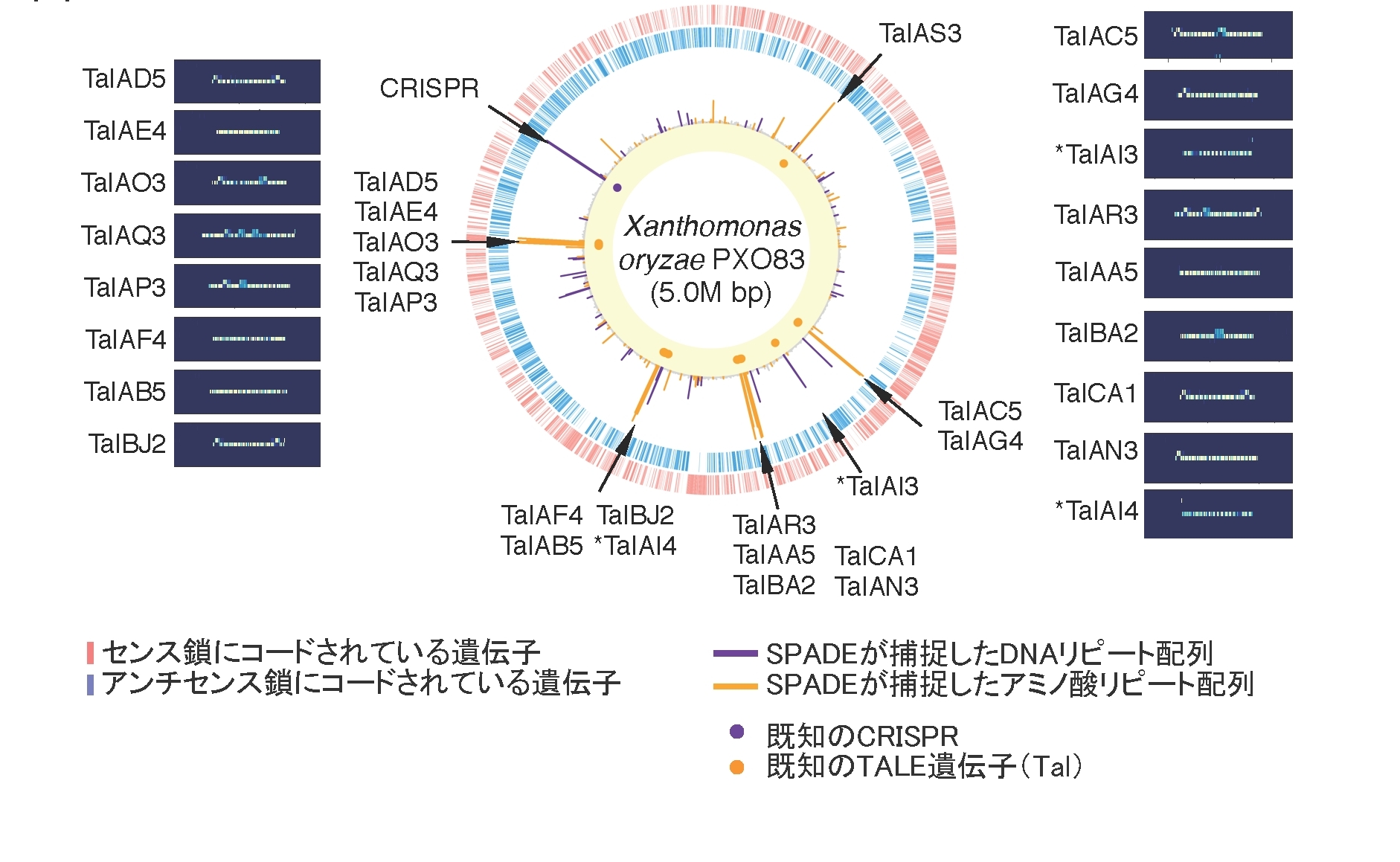
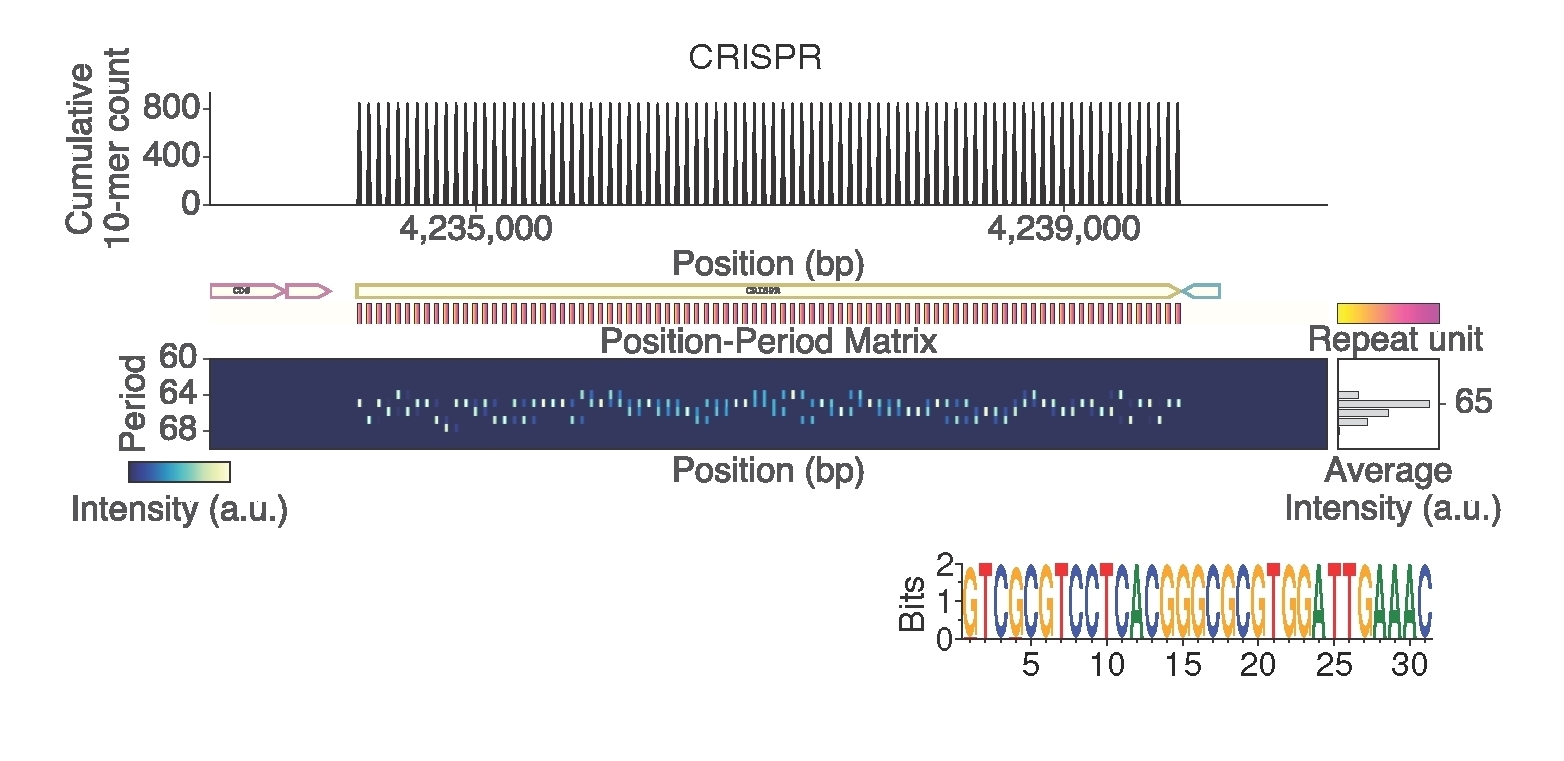
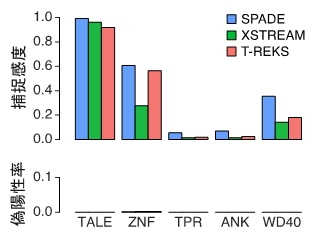
今回開発したSPADEは、大規模なゲノムリソースから高速かつ網羅的に周期的リピート配列を検出することができるソフトウェアです。研究チームはこの新しいソフトウェアを用いて7,006種の微生物ゲノムやヒトゲノムを解析しました。CRISPRやZFN、TALENの由来遺伝子を網羅的に捕捉できるようになった他(図3、図4)、個別にCRISPRやTALEヌクレアーゼの由来遺伝子やその他のリピート配列を予測するためにこれまでに開発されたソフトウェアのどれと比較してもSPADEは同等以上の性能を持つことが示されました(図5)。さらに、SPADEによって新規ゲノム編集関連遺伝子の候補が多く得られ、近年植物から発見され国産ゲノム編集技術としての応用が期待されるPPR(注4)が原核生物にも存在することも示しました。また微生物ゲノム進化の推進に関わったと考えられる周期的リピート配列を多く検出しました。
現在、研究グループは新たな新規ゲノム編集関連遺伝子候補群の検証実験を進めるとともに、SPADEを用いた解析対象を真核生物ゲノムやメタゲノムといった対象に広げています。本ソフトウェアは研究者に向けて公開されており(https://github.com/yachielab/SPADE)、今後生物学において現在利用されているゲノム編集ツールを超えるような新たなゲノム編集ツールの開発や、このような自然界にありながら人工的にも見える規則的な配列から全く新しい生物学的発見が引き起こされることが期待されます。
本研究は国立研究開発法人新エネルギー・産業技術総合開発機構(NEDO)スマートセルプロジェクト「植物等の生物を用いた高機能品生産技術の開発/植物の生産性制御に係る共通基盤技術開発/ゲノム編集の国産技術基盤プラットフォームの確立」の委託業務研究の成果として得られました。
5.発表雑誌:
雑誌名:「Nucleic Acids Research」(オンライン版:10月10日)論文タイトル:Fast and global detection of periodic sequence repeats in large genomic resources
著者:Hideto Mori, Daniel Evans-Yamamoto, Soh Ishiguro, Masaru Tomita, and Nozomu Yachie*
DOI番号:10.1093/nar/gky890
アブストラクトURL:
https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gky890/5124599

6.問い合わせ先:
東京大学先端科学技術研究センター 合成生物学分野准教授 谷内江 望(やちえ のぞむ)
7.用語解説:
(注1)CRISPR原核生物の免疫応答システム。原核生物におけるCRISPRシステムではファージなど外来DNAの感染があると、その外来DNA配列の一部をホストのゲノムに取り込む(免疫)。このホストゲノム領域からは、免疫したDNAと同じ配列をもつRNAが発現し、再び同じ外来DNAの感染があった場合に、このRNAがDNA切断活性をもつCasタンパク質を外来DNAに誘導し、分解する。新たに免疫されるDNA配列は常に一定長で、あるリピート配列に挟まれる形で過去に免疫されたリピート配列の隣に追加される。このため、ゲノム上には一定間隔を空けた周期的なリピート配列が生じ、これがCRISPR(Clustered Regularly Interspaced Short Palindromic Repeats)という名称の由来になった。このメカニズムを利用して、人工的に設計したRNAによってターゲットとなるDNAを自在に切断するという技術がゲノム編集技術として爆発的に利用が広がっている。
(注2)ZFN
転写因子などが持つDNA結合性のドメインをもつジンクフィンガータンパク質を利用したゲノム編集ツール。ジンクフィンガードメインはリピート構造を持ち、DNA二重らせんに巻き付く。各リピート内の一部のアミノ酸の配列パターンが結合するDNA配列に対応しているため、人工的に任意のDNAに結合するジンクフィンガードメインをデザインすることができる。ZFNはこれにDNA切断活性をもつタンパク質ドメインを融合させ、自在に染色体ゲノムを切断できるようにしたもの。
(注3)TALEN
転写因子などが持つDNA結合性のドメインであるTALEを利用したゲノム編集ツール。TALEはリピート構造を持ち、DNA二重らせんに巻き付く。各リピート内の一部のアミノ酸の配列パターンが結合するDNA配列に対応しているため、人工的に任意のDNAに結合するTALEをデザインすることができる。TALENはこれにDNA切断活性をもつタンパク質ドメインを融合させ、自在に染色体ゲノムを切断できるようにしたもの。
(注4)PPR
植物で発見されたRNAおよびDNA結合性のタンパク質。ジンクフィンガーやTALEのようにリピート配列をもち、ジンクフィンガーやTALEのように各リピート内の一部のアミノ酸配列を変化させることで任意のRNAやDNAに結合させることができる。国内でこれを任意のRNAや染色体ゲノムを操作する技術にしようという研究開発が進んでいる。
8.添付資料:
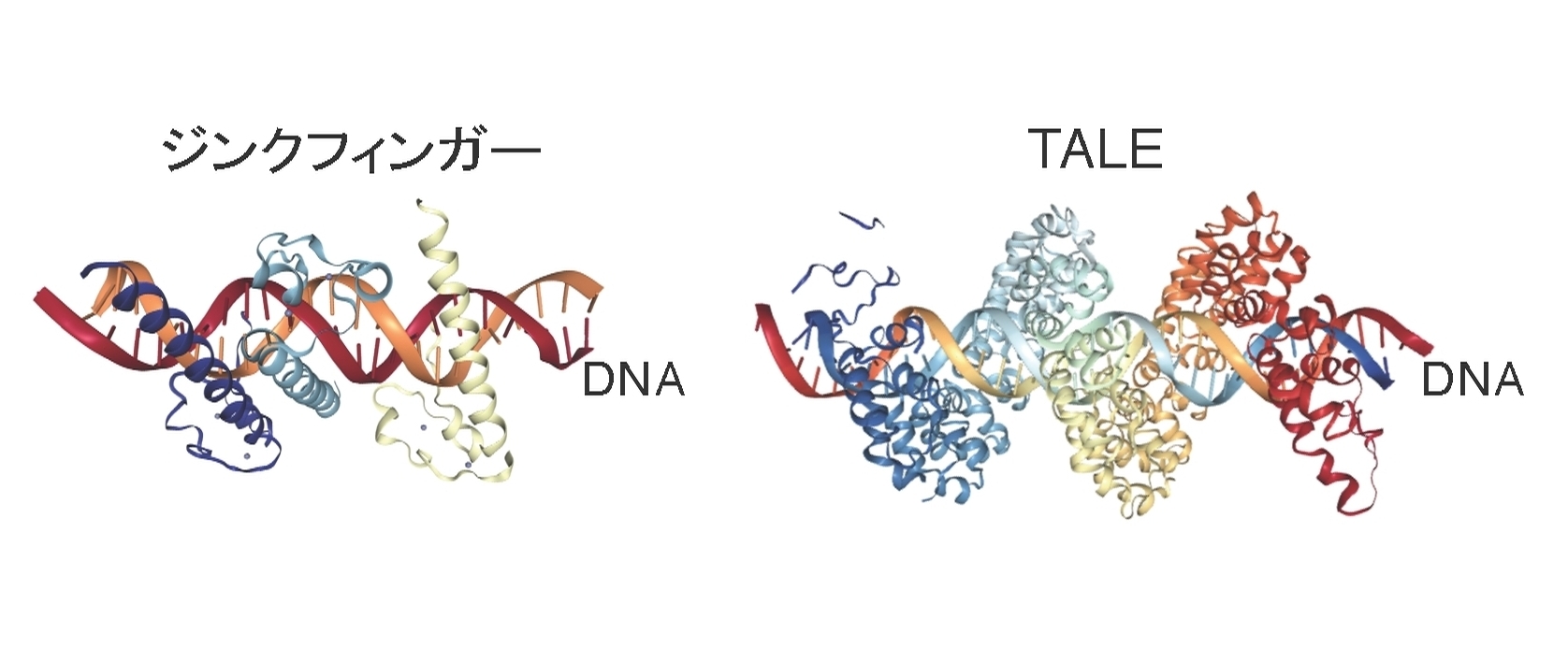
図1:ジンクフィンガードメインやTALEドメインが周期的なタンパク質構造によってDNA二重らせんに巻き付く構造。
関連タグ

