特定の運命を持つ細胞を取り出す技術
―再生医療、細胞進化研究などに幅広く応用―
- 研究成果
2025年5月27日
研究成果のポイント
- 複数の細胞が混ざる細胞集団から特定の運命を持つ細胞だけを単離するCloneSelect法を開発
- 全能性を持った幹細胞に誘導されやすいヒト多能性幹細胞を単離し、解析することに成功
- 幹細胞生物学、再生医療、進化生物学など幅広い分野への貢献に期待
概要
大阪大学ヒューマン・メタバース疾患研究拠点(WPI-PRIMe)創造的破壊生物学分野特任教授、ブリティッシュコロンビア大学(UBC)Biomedical Engineering教授および東京大学先端科学技術研究センター客員教授の谷内江望、UBC School of Biomedical Engineeringの石黒宗博士研究員、同修士課程学生(当時)の坂田莉奈氏、Spiberの石田花菜研究員、京都大学iPS細胞研究所高島康弘教授らの研究グループは、ヒト、マウス、出芽酵母、大腸菌といった様々な細胞集団から特定の運命を辿ることになると考えられる細胞クローンを高精度に取り出す新技術CloneSelect法を開発しました。CloneSelect法が可能にする「遡る生物学」というコンセプトは、細胞の運命をさかのぼって調べるという新しい考え方です。この技術は、これからの発生生物学、幹細胞生物学、再生医療、進化生物学、遺伝子合成といった幅広い分野への貢献が期待されます。本研究成果は、2025年5月21日(水)18時(日本時間)に英国科学誌「Nature Biotechnology」(オンライン)に掲載されました。

ー研究者からのひとことー
本研究は、8年ほど前に緩やかにスタートしました(長い!と思われるかもしれません)。当時CRISPRゲノム編集技術が哺乳類の細胞で可能になった頃で、私が「こういうことができないかな」と研究室のミーティングで発言したところ、当時博士の学生だった石黒君が今の実装を思いついてくれました。以降、Spiber社や京都大学CiRAのすばらしい共同研究者らを得ながら凄いスピードで進んだり、また歩みを緩めて時間のあるときに進めたりしました。休日はしっかり取って、気分の乗ったときにハイスタンダードの仕事をするという石黒君が他の沢山のプロジェクトの合間に良い仕事をしてくれました。また研究室が東京大学からUBCへ移動し、大阪大学でも拠点を構える中で、様々なアプリケーションへの期待とともに、沢山の研究費にサポートして頂きました。まずは地味に見えるかもしれませんが、骨太の技術論文の発表です。そのインパクトはもちろん、研究の進め方の一つとしてよいモデルにもなればいいなと思います。
(東京大学先端科学技術研究センター 客員教授 谷内江望)
研究の背景
細胞が様々な役割に分かれていく「分化」や、生き物の体が形作られる「個体発生」、さらにがんができる「腫瘍形成」など、多くの生命現象は、多様な細胞とそれらの運命が協奏することによって作り出される複雑なシステムです。近年の生物学では、細胞一つ一つの全遺伝子発現情報など生命現象を高度に解析する技術が登場した一方で、これらのデータを得るためには対象の細胞をすり潰し内容物を取り出して破壊的に計測する手法が一般的に取られています。この場合、一度計測した細胞や個体と同じものを異なる時刻で再び調べ、生命現象の動態を調べることはできません。例えば、腫瘍に潜むごく少数の細胞が抗がん剤に対して薬剤耐性を示して増殖し、がん治療を難しくすることが知られています。しかしながら、そのような細胞が初期の腫瘍に最初から存在したのか、あるいは抗がん剤の暴露がそのような細胞の誕生を許してしまったのかなどを調べることは、今日の生物学や医学では容易ではありません。
研究の内容
今回、本研究グループは、CRISPR一塩基ゲノム編集法※1とDNAバーコード法※2を組み合わせることにより、「CloneSelect」と名付けられた新しい遡及的クローン単離法の開発に成功しました。
遡及的クローン単離とは、性質の異なる多くの細胞集団の中から、目的の形質を持つ細胞や、目的の運命を辿る特定の細胞を取り出すことができる新しい技術のコンセプトで、これまでに世界中で様々な技術が開発されています。この技術では、はじめに飛行機の預け入れ荷物にバーコードタグを付けるように、任意の細胞集団の一つ一つの細胞に対して固有のDNAバーコードを安定的に導入します(図1)。次に、このバーコード付きの細胞集団をごく短い期間培養し、それぞれのバーコードを持つクローンを増幅してから2つの集団に分けます。1つの集団を冷凍保存などでストックし、もう一方を薬剤耐性実験や、細胞分化実験などの目的に応じた実験に使います。実験後、「よく増殖する」などの特定の性質を示す細胞集団のDNAバーコードを、超並列DNAシークエンシング※3を用いて解析することで、その集団に多く占められるDNAバーコードを同定することができます。その後、初期に保存しておいた元の細胞集団に戻り、同じDNAバーコードを持つ細胞クローンを蛍光標識して、フローサイトメトリーセルソーティング※4などによって集団から単離することで、目的のクローンを取り出せます。つまり、「将来特定の運命を示すことになる細胞」をその運命が示される以前の細胞集団から取り出して調べることができるのです。今回研究チームが開発したCloneSelect法は様々な点で従来法を大きく上回りました。
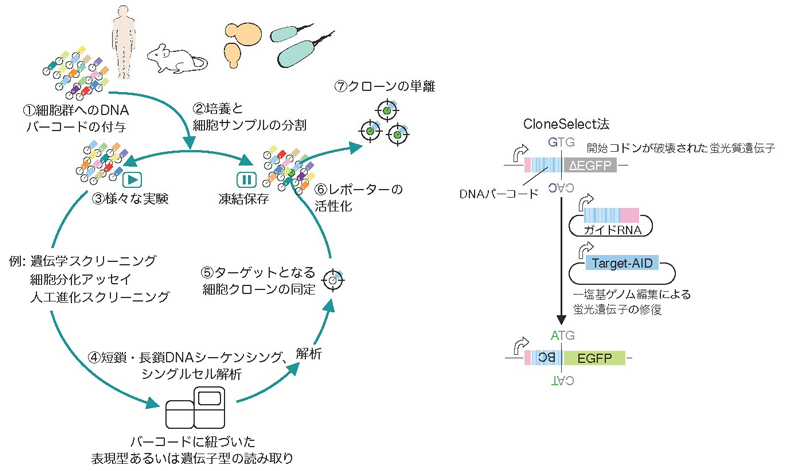
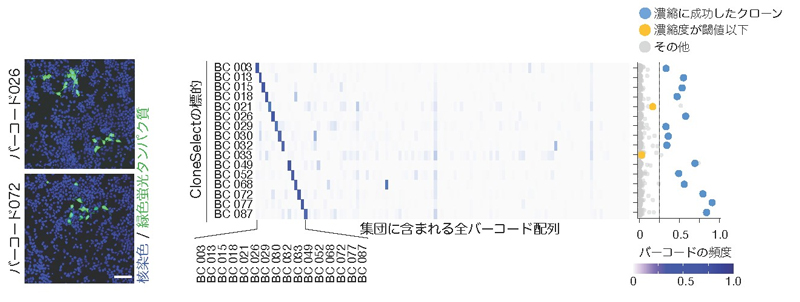
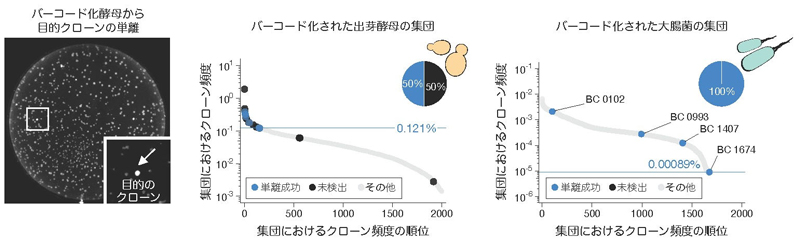
CloneSelect法では、それぞれの細胞を標識するためのDNAバーコードと開始コドンが破壊された緑色蛍光タンパク質遺伝子EGFP (enhanced green fluorescent protein)が隣り合うように配置されます(図1)。このEGFPは通常の状態では発現せず細胞は蛍光を発しません。特定のDNAバーコードを持つ細胞クローンを取り出したい場合には、CRISPR一塩基ゲノム編集法を使って狙ったバーコードの情報に応じてEGFP遺伝子の開始コドンを修復します。これによりターゲットとする細胞だけがEGFPを発現して蛍光を発するようになります。さらに、蛍光を示す細胞はフローサイトメトリーセルソーティングによって効率よく単離することができます。この仕組みがヒト培養細胞で動作するかを試してみると、狙ったDNAバーコードを標的とする細胞を高精度で単離できることが確かめられました(図2)。さらに、この技術はヒトやマウスの幹細胞にも適用可能であることが示されました。また、EGFPを赤色蛍光タンパク質で置き換えて出芽酵母細胞に応用したところ、培養プレート上に形成される大量の細胞コロニー群において、狙ったDNAバーコードを持つクローンだけが蛍光を発し(図3)、爪楊枝などを使って簡単に単離できることを示しました。また、大腸菌では、蛍光タンパク質の代わりに薬剤耐性遺伝子を利用することで、薬剤選択のみでターゲットとなるDNAバーコードを持つ細胞を簡便に得ることができました(図3)。
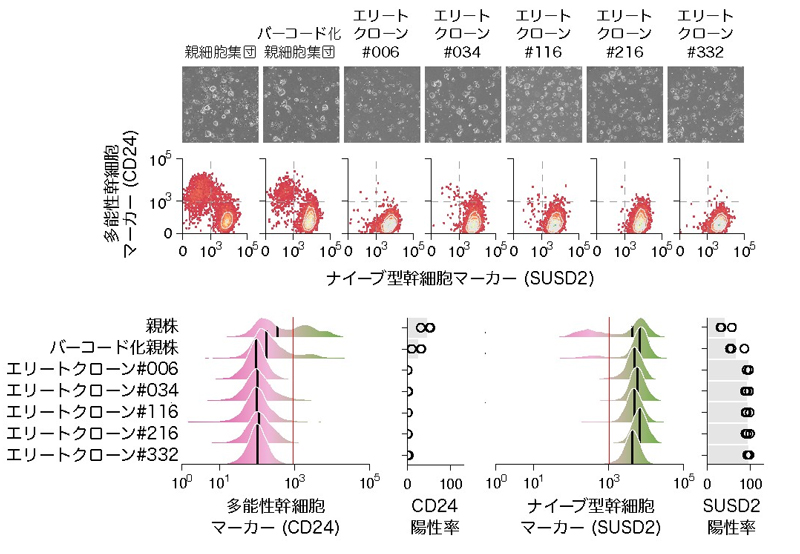
さらに、本研究グループはCloneSelect法をヒト多能性幹細胞からより初期化された状態の幹細胞を誘導するナイーブ化※5実験に応用しました。ヒト多能性幹細胞は、その名の通り、体の様々な細胞に分化するポテンシャルを持つ細胞です。既に、多能性幹細胞がヒト組織を構成するあらゆる細胞への誘導や試験管内での臓器形成実験に応用され始めて久しいですが、それぞれの臓器を形成しやすい多能性幹細胞クローンがあることなども知られていました。各細胞に「細胞分化運命」や「臓器形成運命」があるのではないか?それを解明し、制御する方法は再生医療などに大きく役立つのではないか?と考えられるようになり、同様に、多能性幹細胞はより初期化された状態のナイーブ型の幹細胞へと分化誘導する手法も確立していますが、この誘導効率も100%ではなく、細胞によって誘導のしやすさが異なり、その原因はまだ十分に解明されていませんでした。
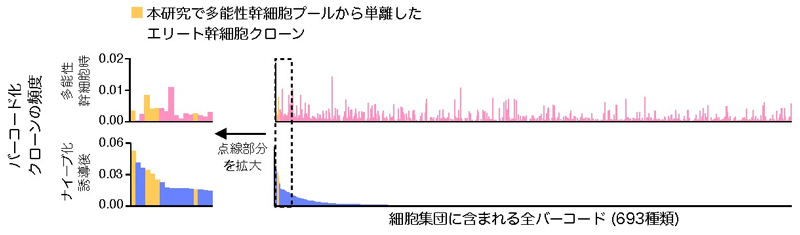
このような中で今回、DNAバーコード化したヒト多能性幹細胞集団をナイーブ化する実験をしたところ、ごく少数の限られたバーコード化クローンのみが安定的かつ高効率にナイーブ型に変化すること(図4)がわかりました。さらに興味深いことに、そのような細胞クローンの運命はある程度培養し続けた多能性幹細胞集団においても変わらないことが示されました。これらの結果から、ナイーブ化誘導されやすいエリートの多能性幹細胞クローンが存在すること、そのような細胞の運命は培養を繰り返してもエリート細胞内になんらかの方法で記録されていることが示唆されました。さらに、いくつか選んだエリートクローンを多能性幹細胞集団から単離して、これらがほぼ完全にナイーブ型幹細胞へと誘導されること(図5)、そのようなエリートクローンは多能性幹細胞の初期状態で特定のエピゲノム※6パターンとそれに規定されるわずかな遺伝子発現の違いを持つことが示唆されました。
本研究成果が社会に与える影響(本研究成果の意義)
生物学にはまだ「複雑な多細胞システムの時系列を容易に解析できない」という課題に根差した多くの未解決問題があります。例えば、上に挙げた抗がん剤に暴露されたがん細胞集団のうち、一部が耐性を獲得し、腫瘍が再発する分子メカニズムはよく分かっていません。また、人体を構成する幹細胞たちがどのような分子に規定されてその発生を制御するのか、損傷した組織を修復するのかも直接観察することができません。進化生物学では、出芽酵母を使った実験室内進化モデルの開発が進んできましたが、未だにどのような変異を獲得することが環境の中で優勢になっていくのかを直接観測したり、再現したりはできません。また大腸菌などを使った遺伝子合成技術の開発も進んでいますが、細胞集団のなかに正しい合成産物遺伝子を持った細胞がわずかしかいない場合は、それを取り出すのが極めて困難なため、そのような細胞サンプルは破棄されています。CloneSelectは、ヒト、マウス、出芽酵母、大腸菌の全てにおいて、生物学的、医学的、産業的に面白い細胞クローンが集団の中に確認された場合に、それを効率よく単離できる方法であり、従来の生物学ではアプローチできなかった生命現象を広く探れるようになる新技術です。これが生物学、医学のコミュニティで利用されるようになることで、幅広い応用が期待されます。
特記事項
本研究成果は、2025年5月21日(水)18時(日本時間)に英国科学誌「ネイチャー・バイオテクノロジー(Nature Biotechnology)」(オンライン)に掲載されました。
- タイトル:
- “A multi-kingdom genetic barcoding system for precise clone isolation”
- 著者名:
- Soh Ishiguro*, Kana Ishida*, Rina C. Sakata*, Minori Ichiraku, Ren Takimoto, Rina Yogo, Yusuke Kijima, Hideto Mori, Mamoru Tanaka, Samuel King, Shoko Tarumoto, Taro Tsujimura, Omar Bashth, Nanami Masuyama, Arman Adel, Hiromi Toyoshima, Motoaki Seki, Ju Hee Oh, Anne-Sophie Archambault, Keiji Nishida, Akihiko Kondo, Satoru Kuhara, Hiroyuki Aburatani, Ramon I. Klein Geltink, Takuya Yamamoto, Nika Shakiba, Yasuhiro Takashima, and Nozomu Yachie** (*共同筆頭著者; **責任著者)
- DOI:
- https://doi.org/10.1038/s41587-025-02649-1
なお本研究は長年に渡り、新エネルギー・産業技術総合開発機構(NEDO)「スマートセルプロジェクト」、科学技術振興機構(JST)戦略的創造研究推進事業さきがけ「1細胞レベルで細胞系譜を一斉同定するDNABarclockテクノロジー」(JPMJPR14FE)、同 CREST「DNAイベントレコーダー細胞」(JPMJCR23B7)、日本学術振興会(JSPS)学術変革B「ゴーギャン生物学」、日本医療研究開発機構(AMED)「機能低下プログラム」、「再生・細胞医療・遺伝子治療実現加速化プログラム」、セコム科学技術振興財団、武田科学振興財団、文部科学省世界トップレベル研究拠点プログラム(WPI)、Canada Foundation for Innovation (CFI)、Canadian Institutes of Health Research (CIHR)、Canadian Institute for Advanced Research (CIFAR) MacMillan Multiscale Human program、Allen Distinguished Investigator Awardからの助成や支援の中で行われました。
用語説明
- ※1 CRISPR一塩基ゲノム編集法
DNA配列の狙った一塩基を置換するゲノム編集手法。CloneSelect法ではDNA配列の狙った位置にあるシトシン(C)をチミン(T)に変換するTarget-AID法とアデニン(A)をグアニン(G)に変換するABE法が用いられる。 - ※2 DNAバーコード法
もともとは、DNAの配列情報を使ってその生物の種名を同定する技術。DNAは20塩基程度の長さになると、420(=1012)種類もの多様性が理論的には生み出せる。近年ではランダムに合成したDNAを使い、一つ一つの細胞を標識したあとに、そのバーコード配列の量を一斉に測って、それぞれの細胞の増殖を見積もるなどといった方法が用いられている。この他にも、様々な生物試料や現象にDNAバーコードを紐づけて、計測の高速化や多重化が実現されている。 - ※3 超並列DNAシークエンシング
ゲノムDNAなど大量のDNAの配列を一斉に解読する方法。DNAバーコードを解析するときは、それぞれのDNAバーコードが何回読み出されるかを数え上げることで、それらを定量することができる。 - ※4 フローサイトメトリーセルソーティング
細胞の大きさや形、さらには細胞が発する蛍光の特徴をもとに、目的の細胞だけを生きたまま高速で選び出すことができる技術。この方法により、多数の細胞が混在するサンプルから、特定の機能や性質をもつ細胞を正確に取り出すことが可能になる。 - ※5 ナイーブ化
ここでは、多能性幹細胞を受精卵近くまで初期化すること。より分化能力が高く、再生医療への応用が期待される。 - ※6 エピゲノム
DNAの塩基配列そのものを変えることなく、遺伝子の働きを制御する仕組みの総称。例えば、DNAのメチル化修飾があり、これは遺伝子の発現を抑える役割を果たす。エピゲノムの特徴は、その情報が細胞分裂のたびにコピーされ、培養条件下でも世代を超えて引き継がれることである。このため、同じDNAをもつ細胞でも異なるエピゲノム状態によって異なる性質を示すことがある。
参考URL
谷内江研究室 https://yachie-lab.org/
問合せ先
東京大学先端科学技術研究センター 客員教授 谷内江 望(やちえ のぞむ)
関連タグ

